Table of Content
- 1 Introduction
- 2 purrr::map() function
- 3 How to request with map() some statistical values
- 4 How to use map() for multiple t-tests
- 5 How to use map() for multiple linear regressions
- 6 Conclusion
library(tidyverse)
library(purrr)
library(broom)
library(knitr)
library(psych)affairs <- read_csv("Affairs.csv")1 Introduction
Sehr häufig werden R-Befehle per Copy & Paste vervielfacht und anschließend für unterschiedliche Zwecke leicht abgewandelt. Dies ist nicht nur umständlich, sondern auch leicht fehleranfällig. Vor diesem Hintergrund werden im Nachfolgenden Möglichkeiten aufgezeigt, wie man effektive Codes in R schreiben kann, ohne einen Befehl doppelt zu verwenden.
Für diesen Post wurde der Datensatz Affairs von der Statistik Plattform “Kaggle” verwendet. Eine Kopie des Datensatzes ist unter https://drive.google.com/open?id=1N4osROEo724c7OZA2ARiwEthcZDwLxtf abrufbar.
2 purrr::map() function
Als kurze Einführung wird die Frage beantwortet, was die map Funktion aus dem purrr Package macht.
v <- list(1, 2, 3)
map(v, ~ .* 10)## [[1]]
## [1] 10
##
## [[2]]
## [1] 20
##
## [[3]]
## [1] 30Die map Funktion transformiert einen Input durch die Anwendung einer Funktion auf jedes Element des Inputs und gibt anschließend einen Vektor aus, der dieselbe Länge besitzt wie die des Inputs. In unserem Beispiel wurde R der Befehl gegeben, jedes Element aus v (zuvor erstelltes Objekt) mit 10 zu multiplizieren.
3 How to request with map() some statistical values
glimpse(affairs)## Observations: 601
## Variables: 10
## $ X1 <int> 4, 5, 11, 16, 23, 29, 44, 45, 47, 49, 50, 55, 64...
## $ affairs <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, ...
## $ gender <chr> "male", "female", "female", "male", "male", "fem...
## $ age <dbl> 37, 27, 32, 57, 22, 32, 22, 57, 32, 22, 37, 27, ...
## $ yearsmarried <dbl> 10.00, 4.00, 15.00, 15.00, 0.75, 1.50, 0.75, 15....
## $ children <chr> "no", "no", "yes", "yes", "no", "no", "no", "yes...
## $ religiousness <int> 3, 4, 1, 5, 2, 2, 2, 2, 4, 4, 2, 4, 5, 2, 4, 1, ...
## $ education <int> 18, 14, 12, 18, 17, 17, 12, 14, 16, 14, 20, 18, ...
## $ occupation <int> 7, 6, 1, 6, 6, 5, 1, 4, 1, 4, 7, 6, 6, 5, 5, 5, ...
## $ rating <int> 4, 4, 4, 5, 3, 5, 3, 4, 2, 5, 2, 4, 4, 4, 4, 5, ...Für die Beschreibung eines Datensatzes werden häufig sehr viele statistische Kennzahlen (Median, Mittelwert, Standardabweichung, …) angefordert. Dies jeweils mit einem eigenen Befehl zu tun ist besonders bei großen Datensätzen mit vielen Variablen sehr zeitintensiv und auch völlig überflüssig, denn hierfür kann prima auch die map Funktion verwendet werden.
Zu Beginn einer Analyse ist meist von Interesse, ob und wenn ja wie viele fehlende Werte im Datensatz vorhanden sind. Dies kann folgendermaßen überprüft werden.
affairs %>%
map(~sum(is.na(.)))## $X1
## [1] 0
##
## $affairs
## [1] 0
##
## $gender
## [1] 0
##
## $age
## [1] 0
##
## $yearsmarried
## [1] 0
##
## $children
## [1] 0
##
## $religiousness
## [1] 0
##
## $education
## [1] 0
##
## $occupation
## [1] 0
##
## $rating
## [1] 0Man sieht, dass bei keiner Variablen des Datensatzes “affairs” ein fehlender Wert (NA) existiert. Dieselbe Überprüfung kann auch pro Zeile erfolgen. Aus Übersichtsgründen wurde die Ausgabe des Befehls hier unterdrückt.
apply(affairs,MARGIN = 1, FUN = function(x) sum(is.na(x)))Mit dem select Befehl können auch gezielt Spalten ausgewählt werden, für die anschließend mit der map Funktion die Kennzahlen angefordert werden sollen.
affairs %>% select(7:10) %>% map(describeBy)## $religiousness
## vars n mean sd median trimmed mad min max range skew kurtosis
## X1 1 601 3.12 1.17 3 3.12 1.48 1 5 4 -0.09 -1.02
## se
## X1 0.05
##
## $education
## vars n mean sd median trimmed mad min max range skew kurtosis se
## X1 1 601 16.17 2.4 16 16.21 2.97 9 20 11 -0.25 -0.32 0.1
##
## $occupation
## vars n mean sd median trimmed mad min max range skew kurtosis
## X1 1 601 4.19 1.82 5 4.34 1.48 1 7 6 -0.74 -0.79
## se
## X1 0.07
##
## $rating
## vars n mean sd median trimmed mad min max range skew kurtosis se
## X1 1 601 3.93 1.1 4 4.07 1.48 1 5 4 -0.83 -0.22 0.04Meistens sind jedoch nur gezielte Kennzahlen von bestimmten Variablen von Interesse. Der Datensatz Affairs wird deshalb auf folgende 5 Variablen beschränkt.
affairs.short <- affairs %>% select(age, 7:10)
affairs.short## # A tibble: 601 x 5
## age religiousness education occupation rating
## <dbl> <int> <int> <int> <int>
## 1 37 3 18 7 4
## 2 27 4 14 6 4
## 3 32 1 12 1 4
## 4 57 5 18 6 5
## 5 22 2 17 6 3
## 6 32 2 17 5 5
## 7 22 2 12 1 3
## 8 57 2 14 4 4
## 9 32 4 16 1 2
## 10 22 4 14 4 5
## # ... with 591 more rowsEs soll nun der Mittelwert für alle 5 Variablen angefordert werden. Dafür würde sich die for loop Funktion anbieten.
output <- vector("double", length(affairs.short))
for (i in seq_along(affairs.short)) {
output[[i]] <- mean(affairs.short[[i]])
}
output## [1] 32.487521 3.116473 16.166389 4.194676 3.931780Man könnte dies auch gleich als Funktion schreiben.
col_mean <- function(df) {
output <- vector("double", length(df))
for (i in seq_along(df)) {
output[i] <- mean(df[[i]])
}
output
}
col_mean(affairs.short) ## [1] 32.487521 3.116473 16.166389 4.194676 3.931780Man sieht, dass die Funktion klappt. Leicht abgewandelt, kann diese auch andere statistische Kennwerte berechnen. Im Nachfolgenden werden die Befehle auf die gleiche Art und Weise für die Standardabweichung und den Median erstellt.
col_sd <- function(df) {
output <- vector("double", length(df))
for (i in seq_along(df)) {
output[i] <- sd(df[[i]])
}
output
}
col_median <- function(df) {
output <- vector("double", length(df))
for (i in seq_along(df)) {
output[i] <- median(df[[i]])
}
output
}Anschließend erfolgt eine gemeinsame Ausgabe der statistischen Werte.
col_mean(affairs.short) ## [1] 32.487521 3.116473 16.166389 4.194676 3.931780col_sd(affairs.short) ## [1] 9.288762 1.167509 2.402555 1.819443 1.103179col_median(affairs.short)## [1] 32 3 16 5 4Auch dies funktioniert wie erwartet. Für eine bessere Übersicht, können die Zeilen miteinander verbunden werden.
Mittelwert<-col_mean(affairs.short)
Median<-col_median(affairs.short)
standardabweichung<-col_sd(affairs.short)
cbind(Mittelwert,Median ,standardabweichung)## Mittelwert Median standardabweichung
## [1,] 32.487521 32 9.288762
## [2,] 3.116473 3 1.167509
## [3,] 16.166389 16 2.402555
## [4,] 4.194676 5 1.819443
## [5,] 3.931780 4 1.103179An und für sich ein zufriedenstellendes Ergebnis. Jedoch wurde wieder ein Fehler gemacht, der in diesem Post eigentlich vermieden werden sollte. Es wurde erneut copy & paste angewendet.
Die zentrale Frage ist: wie können die drei Funktionen in einer Syntax miteinander verbunden werden?
Zur Beantwortug dieser Frage werden beispielhaft folgende drei Funktionen erstellt.
fun1 <- function(x) abs(x - mean(x))^1
fun2 <- function(x) abs(x - mean(x))^2
fun3 <- function(x) abs(x - mean(x))^3Diese in einer Syntax zu schreiben könnte folgendermaßen aussehen:
fun.ges <- function(x, i) abs(x - mean(x))^iGenau dieses Prinzip lässt sich auch auf die Funktionen der statistischen Kennwerte anwenden.
col_summary <- function(df, fun) {
out <- vector("double", length(df))
for (i in seq_along(df)) {
out[[i]] <- fun(df[[i]])
}
out
} Die erstellte col_summary Funktion lässt sich nun beliebig auf jeden Datensatz (Argument 1) und R-Befehl (Argument 2) anwenden. Hier die Anforderung des Medians für die Variablen im Datensatz “affairs.short”
col_summary(affairs.short, median)## [1] 32 3 16 5 4Man sieht das gleiche Ergebnis wie bei der zuvor erstellten “Einzelfunkton” für den Median (col_median(affairs.short)).
4 How to use map() for multiple t-tests
Mit map lässt sich ein kompletter Datensatz auch sehr schnell und effizient auf beispielsweise Geschlechterunterschiede mittels t-tests untersuchen.
affairs %>% select(age, affairs, yearsmarried, education, religiousness) %>%
map(function(x) t.test(x ~ affairs$gender)$p.value)## $age
## [1] 2.848452e-06
##
## $affairs
## [1] 0.7739606
##
## $yearsmarried
## [1] 0.458246
##
## $education
## [1] 9.772643e-24
##
## $religiousness
## [1] 0.8513998Männer und Frauen unterscheiden sich also signifikant in dieser Stichprobe nur in ihrem Alter und der Ausbildung.
affairs %>% select(age, affairs, yearsmarried, education, religiousness) %>% map(function(x) t.test(x ~ affairs$gender)$p.value) %>%
as.data.frame %>% gather %>% mutate(signif = ifelse(value < .05, "significant", "n.significant")) %>%
ggplot(aes(x = reorder(key, value), y = value)) +
geom_point(aes(color=signif)) +
coord_flip() +
ylab("p-value")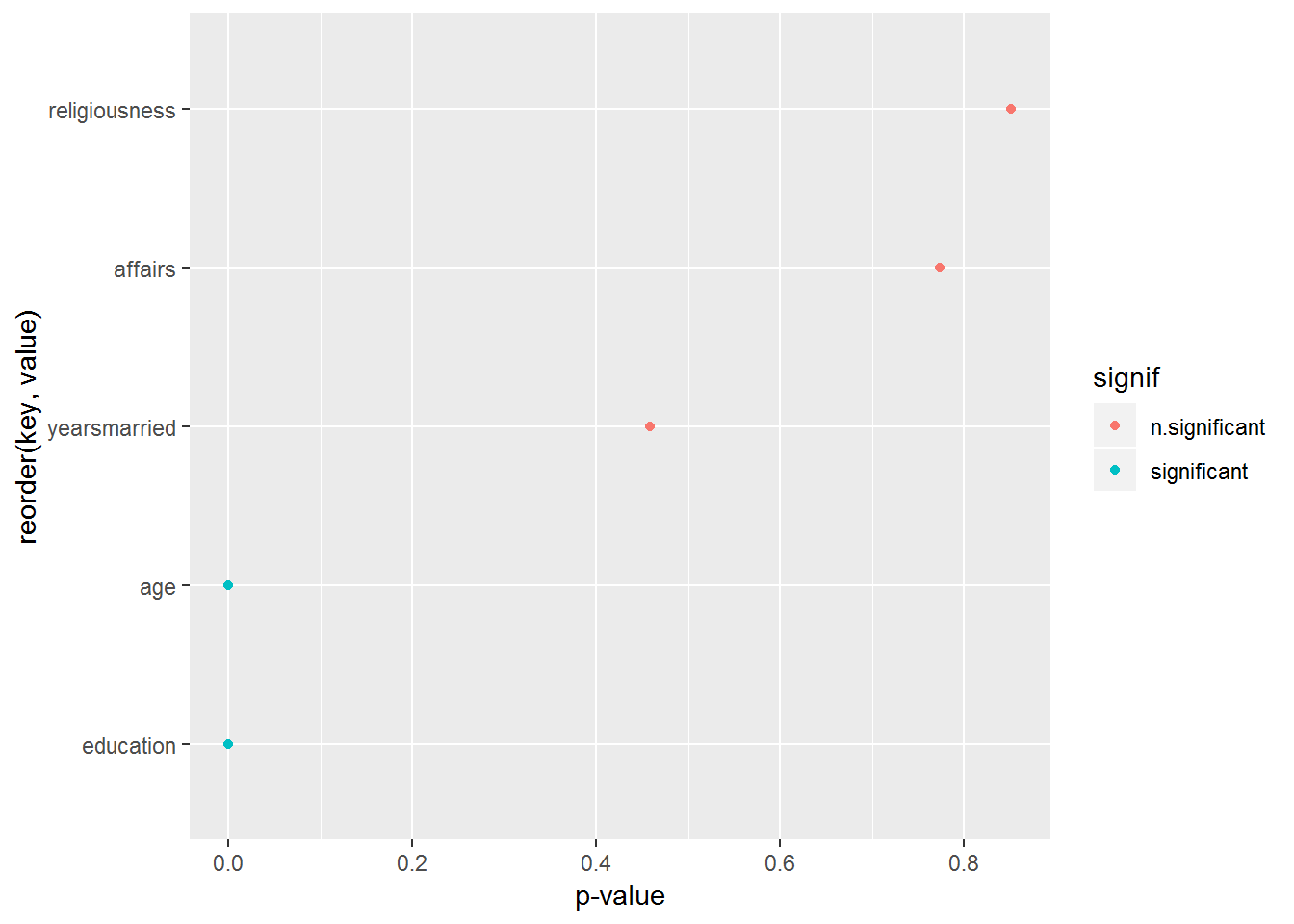
5 How to use map() for multiple linear regressions
Was mit dem t-Test ging, geht auch für die einfache lineare Regression. Hierbei wird aber nicht der p-Wert angefordert, sondern R2.
affairs %>%
select(age, yearsmarried, education, religiousness, rating, children, gender, occupation) %>%
map(~lm(affairs ~ .x, data = affairs)) %>%
map(summary) %>%
map_dbl("r.squared") %>%
tidy %>%
arrange(desc(x)) %>%
rename(r.squared = x) -> modell## Warning: 'tidy.numeric' ist veraltet.
## Siehe help("Deprecated")kable(modell)| names | r.squared |
|---|---|
| rating | 0.0781272 |
| yearsmarried | 0.0349098 |
| religiousness | 0.0208806 |
| children | 0.0108181 |
| age | 0.0090701 |
| occupation | 0.0024613 |
| gender | 0.0001377 |
| education | 0.0000059 |
Der map Befehl sieht hier anders aus, als der beim t-Test verwendete, ist aber nur eine andere Schreibweise. Hier die beiden Varianten gegenüber gesellt:
- map(~lm(affairs ~ .x, data = affairs))
- map(function(x) lm(affairs$affairs ~ .x))
Zur Überprüfung wird eine Regression manuell durchgeführt.
lm1 <- lm(affairs ~ rating, data = affairs)
summary(lm1)$r.squared## [1] 0.07812718Wir sehen das gleiche R2 wie bei der Ausgabe kable(modell).
Wie viele Regressionen wurden eigentlich durchgeführt?
ncol(affairs %>%
select(age, yearsmarried, education, religiousness, rating, children, gender, occupation))## [1] 8Insgesamt 8 Stück. Es hat sich also bereits gelohnt die map Funktion anzuwenden. Die Ergebnisse sollen nun noch schön graphisch dargestellt werden.
ggplot(modell, aes(x = reorder(names, r.squared), y = r.squared)) +
geom_point(size = 5, color = "green") +
ylab(expression(R^{2})) +
xlab("Prädiktoren") +
ggtitle("Erklärte Varianz pro Prädiktor")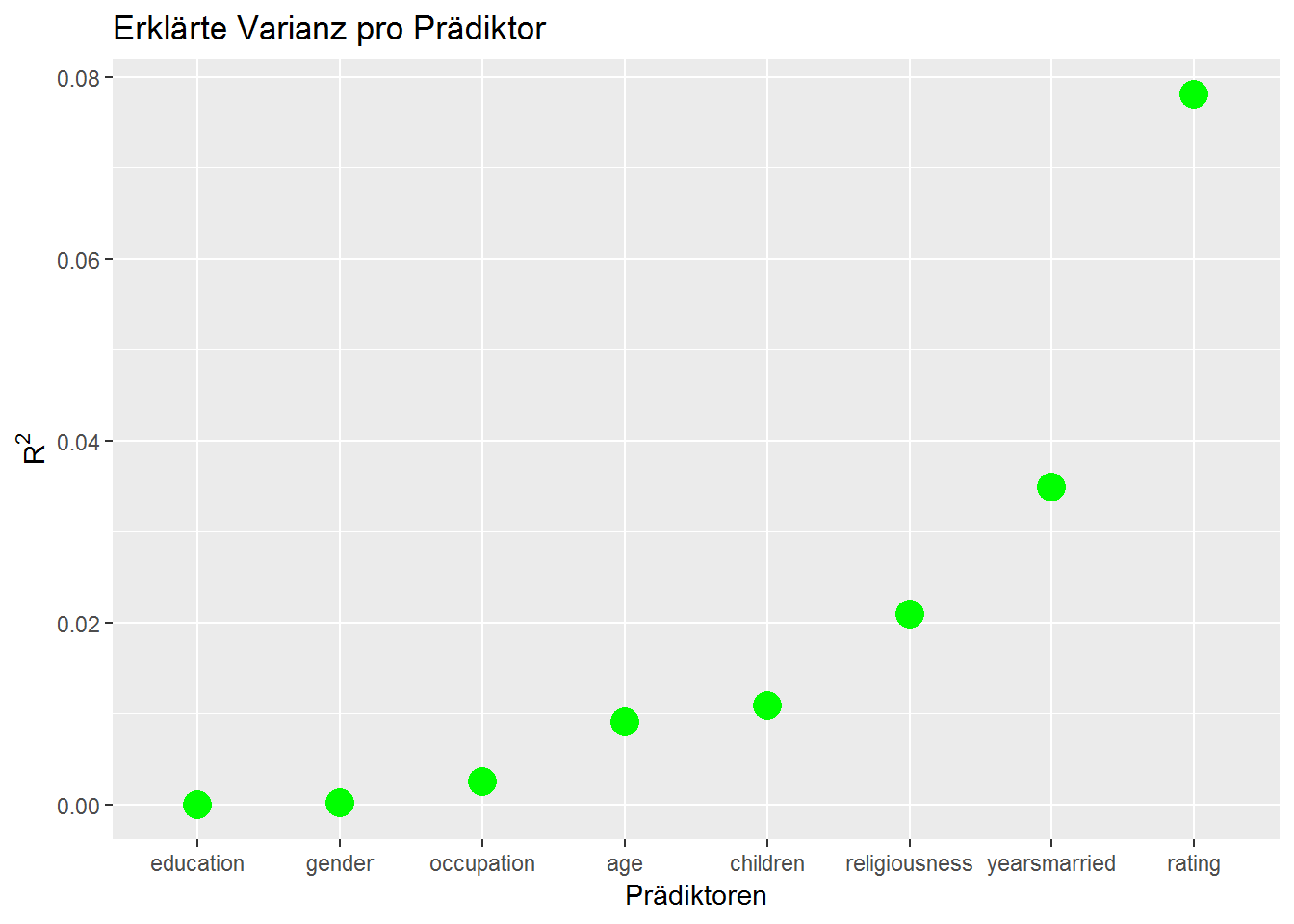
Der Prädiktor rating erklärt also mit knapp 8% am meisten Varianz.
Im zweiten Schritt soll noch überprüft werden, ob die Ergebnisse signifikant sind.
affairs %>%
select(age, yearsmarried, education, religiousness, rating, children, gender, occupation) %>%
map(~lm(affairs ~ .x, data = affairs)) %>%
map(summary) %>%
map(c("coefficients")) %>%
map_dbl(8) %>%
tidy %>%
arrange(desc(x)) %>%
rename(p.value = x) -> modell.pvalue## Warning: 'tidy.numeric' ist veraltet.
## Siehe help("Deprecated")kable(modell.pvalue)| names | p.value |
|---|---|
| education | 0.9524501 |
| gender | 0.7740138 |
| occupation | 0.2245709 |
| age | 0.0195320 |
| children | 0.0107275 |
| religiousness | 0.0003797 |
| yearsmarried | 0.0000040 |
| rating | 0.0000000 |
Eine Gegenüberstellung der Ergebnisse kann folgendermaßen aussehen:
cbind(modell, modell.pvalue)## names r.squared names p.value
## 1 rating 7.812718e-02 education 9.524501e-01
## 2 yearsmarried 3.490982e-02 gender 7.740138e-01
## 3 religiousness 2.088064e-02 occupation 2.245709e-01
## 4 children 1.081809e-02 age 1.953203e-02
## 5 age 9.070125e-03 children 1.072749e-02
## 6 occupation 2.461327e-03 religiousness 3.797263e-04
## 7 gender 1.377396e-04 yearsmarried 3.995520e-06
## 8 education 5.941117e-06 rating 3.002385e-12Dies ist allerdings sehr unschön, da die Reihenfolge der beiden Listen mit absteigenden R2 bzw p-Wert angefordert wurden und daher unterschiedlich angeordnet sind. Abhilfe leistet hier der left_join Befehl.
Gute_Ansicht <- modell %>% left_join(modell.pvalue, by = "names") %>%
mutate(P_Wert = round(p.value, digits = 2)) %>%
select(-p.value)
Gute_Ansicht## # A tibble: 8 x 3
## names r.squared P_Wert
## <chr> <dbl> <dbl>
## 1 rating 0.0781 0
## 2 yearsmarried 0.0349 0
## 3 religiousness 0.0209 0
## 4 children 0.0108 0.01
## 5 age 0.00907 0.02
## 6 occupation 0.00246 0.22
## 7 gender 0.000138 0.77
## 8 education 0.00000594 0.956 Conclusion
Die Verwendung der map Funktion ist mit ein wenig Übung leicht anzuwenden und unglaublich effektiv, wenn es um automatisierte Datenanalyse geht.